Séquençage et analyse de métagénomes : amplicons (16S, 18S, ITS) ou whole genome
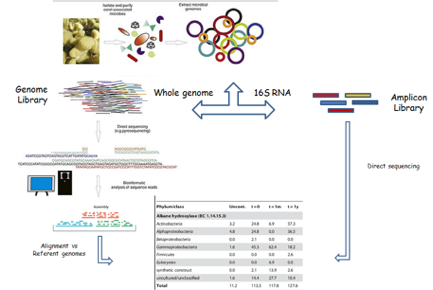
Viroscan3D propose des prestations de séquençage et d’analyse de métagénomes par amplicons (16S, 18S, ITS), Rad-Seq ou whole genome.
Ces prestations visent à étudier le contenu génétique (ADN) d’un échantillon issu d’un environnement complexe clinique (Expectorations, Lavages broncho-alvéolaires) ou environnemental (océan, sols, air), etc.
Nous proposons un pipeline complet de séquençage : du design de votre projet jusqu’à l’analyse bio-informatique des données. Nous vous garantissons un accompagnement absolu de vos projets, la meilleure stratégie d’analyse : nécessité ou non d’enrichir préalablement la population à analyser, meilleure stratégie d’amplification, couverture, profondeur et coût, stratégie d’analyse de données et ce quelque-soit l’échantillon : de mauvaise qualité ou en faible quantité.
Nous vous garantissons la meilleure décision pour le meilleur résultat.
Séquençage et analyse du métagénome
Nature des échantillons traités
Acides nucléiques totaux provenant d’échantillons cliniques ou environnementaux.
Services
- Aide au plan expérimental
- Extraction des acides nucléiques
- Construction des librairies pour séquençage court-fragments et/ou longs fragments, quantification et validation des librairies, séquençage courts fragments Illumina en paired-end et/ou longs fragments Nanopore MinION.
- Analyse primaire des données : génération des fichiers fastq et démultiplexage
- Analyse secondaire des données : liste des agents présents (souche ou espèce en fonction de l’approche)
Livrables
- Rapport d’analyse
- Données brutes de séquençage
- Tableaux des résultats des agents bactériens, viraux, fungiques et autres présents
